scikit-learnの決定木は、どんな条件に木構造がつくられたか、確認できるみたいだね。
条件が確認できると、人間側としてもアルゴリズムが何をしているか、ブラックボックスにならなくていいね。
前回は、決定木について概要を勉強し、どんなふうに分類されていくのか、境界線を見て楽しみました。決定木は、条件を作成して木構造を作るのですが、その内容も確認できることがわかりました。木構造が確認できると、どこでどの条件が不要なのかなど、チューニングするときの一つの判断材料となりそうです。
決定木については前回の記事もご参考ください。

こんな人の役に立つかも
・機械学習プログラミングを勉強している人
・決定木について勉強している人
・scikit-learnの決定木の木構造を表示したい人
決定木を可視化してみる
最初の3ブロック分のコードは、前回記事と同様の内容となっています。データは、scikit-learnの乳がんデータです。
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.model_selection import cross_val_score
from sklearn import tree
import matplotlib.pyplot as plt
import numpy as np
#乳がんデータ
panda_box = load_breast_cancer()
#2個分の特徴量に絞る
X = panda_box.data[:,0:2]
y = panda_box.target
print(panda_box.feature_names)
#訓練データとテストデータに分割
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.25, stratify=y, random_state=0)
#決定木
clf = tree.DecisionTreeClassifier(max_depth=3).fit(X_train,y_train)#関数を作成
def make_meshgrid(x, y, h=.02):
x_min, x_max = x.min() - 1, x.max() + 1
y_min, y_max = y.min() - 1, y.max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, h),
np.arange(y_min, y_max, h))
return xx, yy
def plot_contours(ax, clf, xx, yy, **params):
Z = clf.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
out = ax.contourf(xx, yy, Z, **params)
return out#決定木のグラフ
fig = plt.figure(figsize=(8, 8))
ax = fig.add_subplot(111)
#グリッドのデータを作成
X0 , X1 = X_train[:,0], X_train[:,1]
xx, yy = make_meshgrid(X0, X1)
#グラフに境界線とデータをプロット
plot_contours(ax, clf, xx, yy, cmap=plt.cm.coolwarm, alpha=0.8)
ax.scatter(X0, X1, c=y_train, cmap=plt.cm.coolwarm, s=20, edgecolors='k')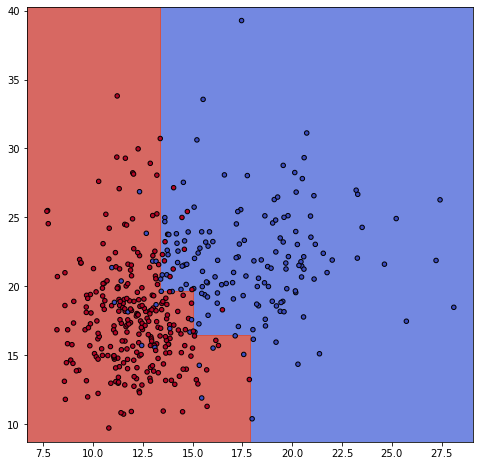
次に決定木の内容を確認します。
plot_treeは、axのパラメータにmatplotlibの描画領域を渡すことで画像サイズを整えることができます。
#決定木の中身を確認
#matplotlibで描画領域の作成
fig_dt = plt.figure(figsize=(16,8))
ax_dt = fig_dt.add_subplot(111)
#木構造を出力
tree.plot_tree(clf, fontsize=10.5, ax = ax_dt)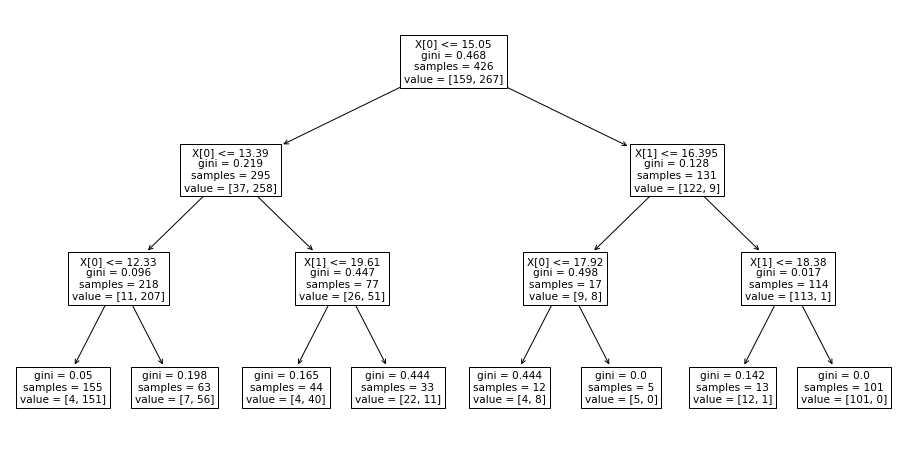
木構造の機能が追加されたのが最近なので、Web検索では、GraphVizという外部の機能を利用して描くサンプルが多いです。scikit-learn0.21からは簡単に出力できるようになりました。
最初はgraphVizというライブラリで保存したり読み出したり、めんどいなと思ってました。
パラメータの調整
木構造の条件を追うことで、「ここの条件が細かすぎる」などの分析ができ、決定木のパラメータを調整することができます。
今までの条件は、下のように深さ3の決定木を作成していました。
DecisionTreeClassifier(max_depth=3)その時のテストデータへの精度も次のようになっています。
テストデータへの精度
0.8741パラメータを色々と触ってみましたが、一番効果があるのが、深さを変更することでした。
それ以外のパラメータはあまり理解できていませんが・・・
ということで、max_depthが4の時は、次のようになりました。
DecisionTreeClassifier(criterion='gini',max_depth=4)テストデータへの精度
0.8881
右に細く伸びているところは気になるよね、汎化性能が高い境界線には思えないよ。
今回は、二次元の特徴量なので、パラメータでどうにかするというよりも、特徴量の数を増やす方が良いのかもしれません。
ちなみに、乳がんデータ全ての特徴量を入れた時のテストデータの精度はこのようになりました。
テストデータへの精度
0.9231
